Mapa de calor con dendrogramas con clustermap
La función clustermap es muy similar a la función heatmap. La principal diferencia es que clustermap también calculará y representará la agrupación jerárquica de las filas y columnas de los datos.
import numpy as np
import seaborn as sns
# Simulación de datos
np.random.seed(2)
data = np.random.rand(6, 6)
sns.clustermap(data)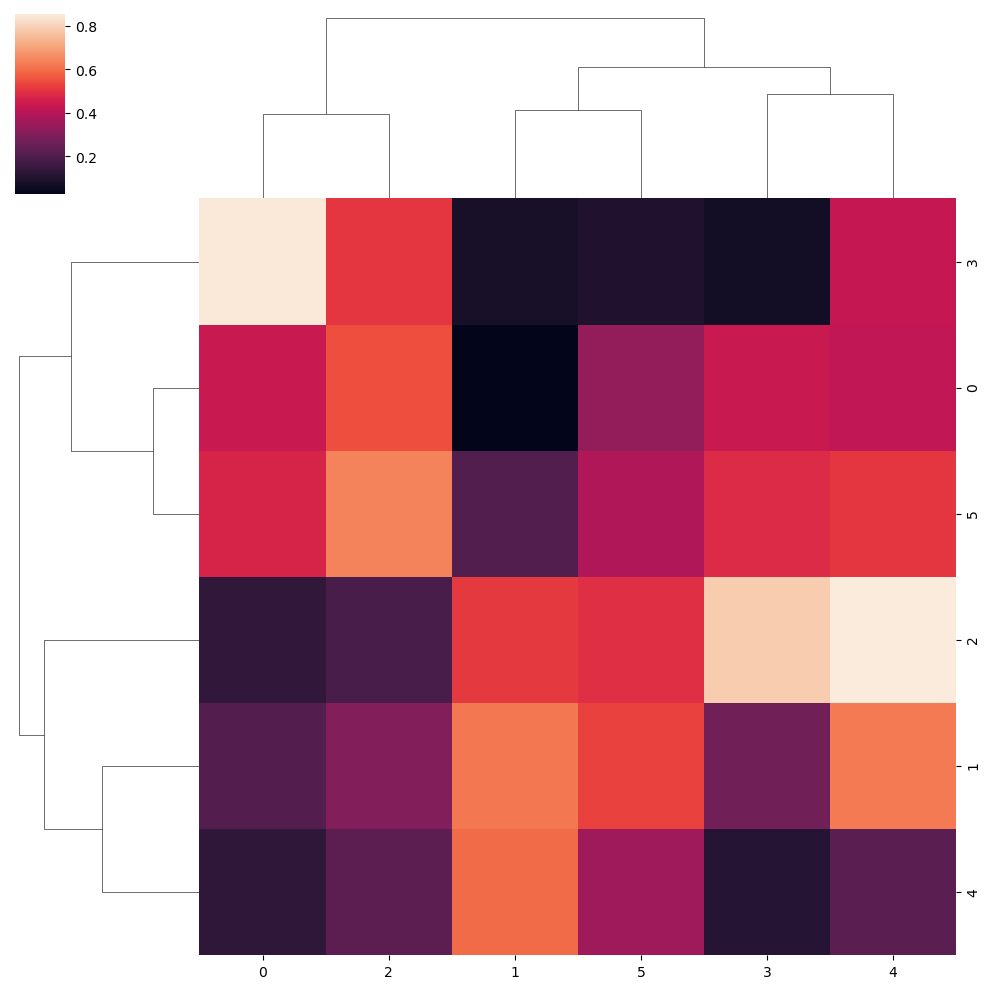
Datos estandarizados
Ten en cuenta que puedes estandarizar los datos por filas (0) o por columnas (1) con el argumento standard_scale de la función.
import numpy as np
import seaborn as sns
# Simulación de datos
np.random.seed(2)
data = np.random.rand(6, 6)
sns.clustermap(data, standard_scale = 1)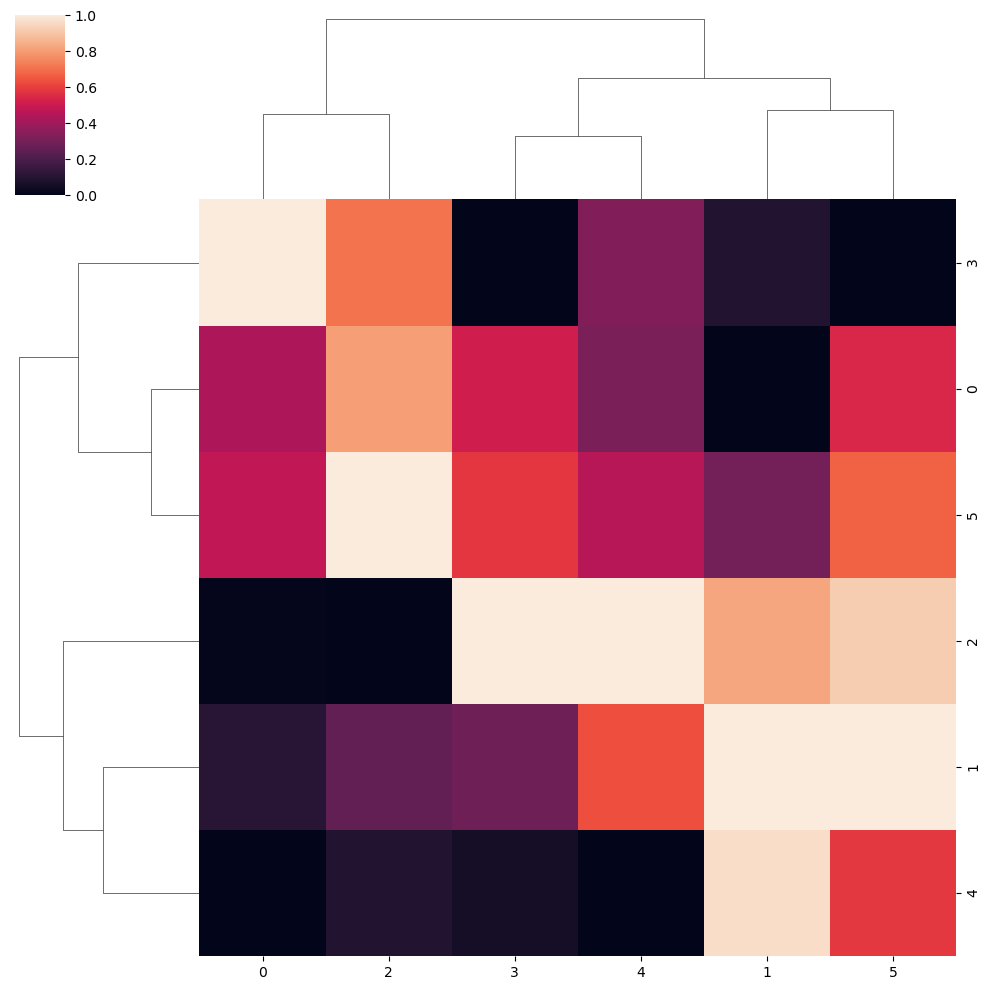
Datos normalizados
De manera similar, también es posible normalizar los datos por filas (0) o por columna (1) con el argumento z_score.
import numpy as np
import seaborn as sns
# Simulación de datos
np.random.seed(2)
data = np.random.rand(6, 6)
sns.clustermap(data, z_score = 1)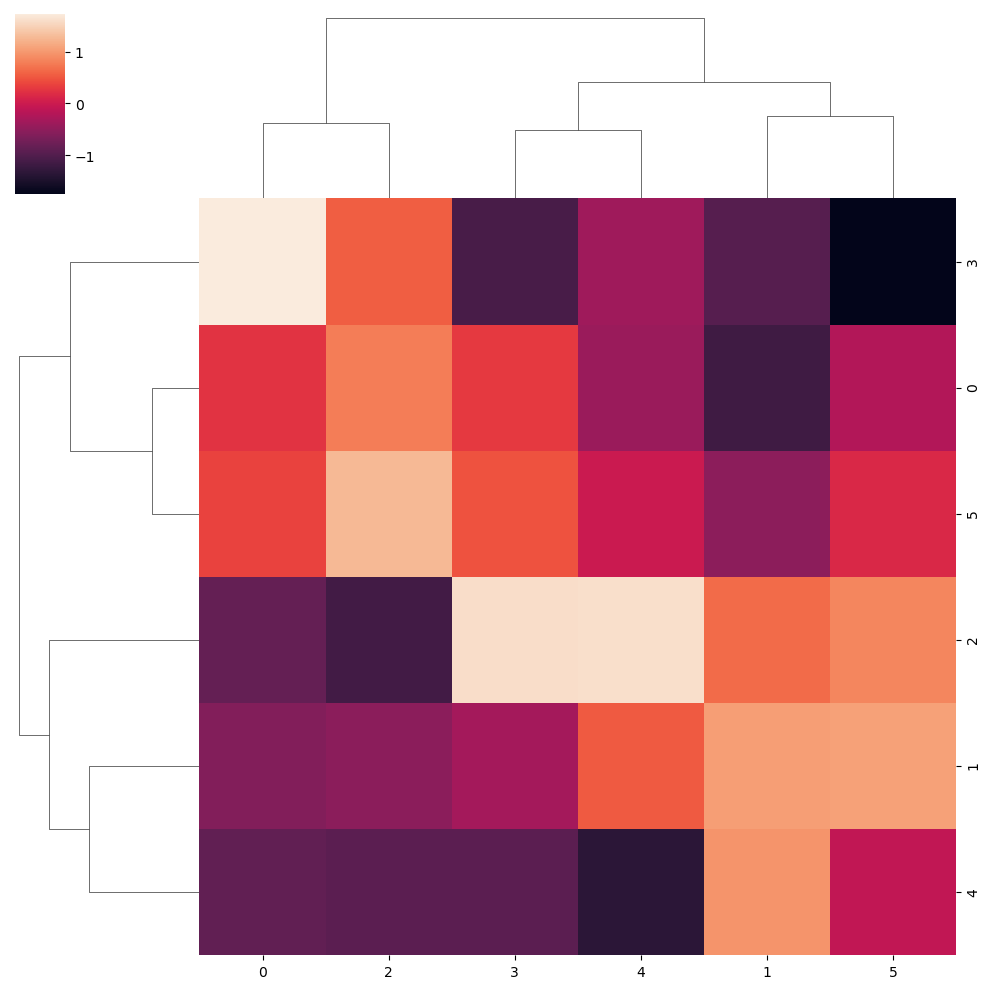
Tamaño de la figura
Ten en cuenta que puedes personalizar el tamaño de la figura pasando un vector de tamaños a figsize, donde el primero representa el grosor y el segundo la altura de la figura. Además, puedes establecer el tamaño relativo del dendrograma con respecto al tamaño total del gráfico con dendrogram_ratio.
import numpy as np
import seaborn as sns
# Simulación de datos
np.random.seed(2)
data = np.random.rand(6, 6)
sns.clustermap(data,
figsize = (8, 6), # Tamaño de la figura
dendrogram_ratio = 0.1) # Proporción de tamaño de los dendrogramas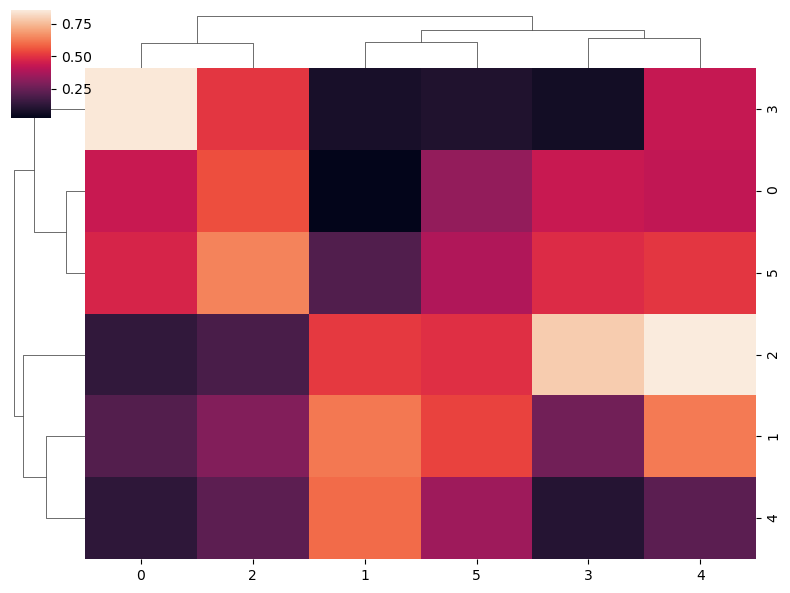
Método de clustering
El método usado para calcular los clústers jerárquicos se puede seleccionar con el argumento method. Las posibles opciones son: "single", "complete", "average" (por defecto) "weighted", "centroid", "median" y "ward". En los siguientes bloques de código puedes ver un par de ejemplos de uso. Revisa esta referencia para obtener detalles adicionales sobre cada método.
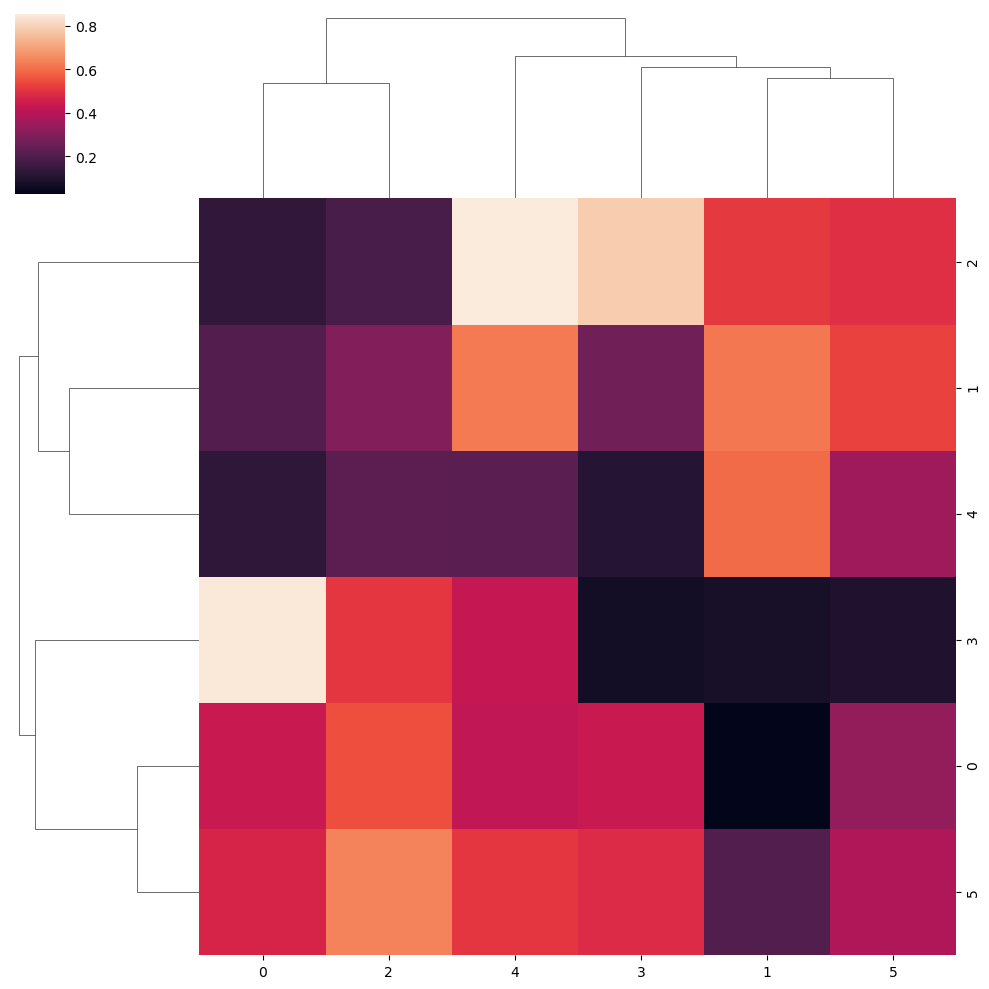
Método “single”
import numpy as np
import seaborn as sns
# Simulación de datos
np.random.seed(2)
data = np.random.rand(6, 6)
sns.clustermap(data, method = "single")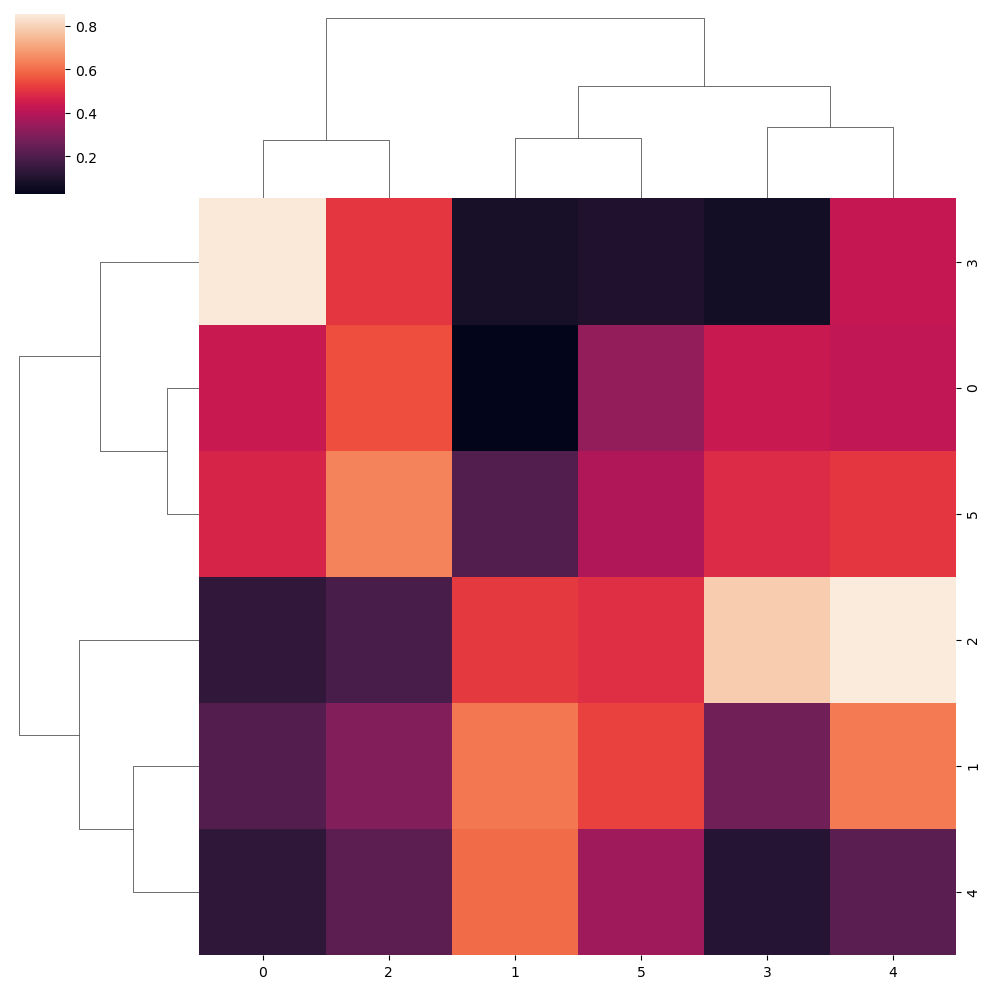
Método “ward”
import numpy as np
import seaborn as sns
# Simulación de datos
np.random.seed(2)
data = np.random.rand(6, 6)
sns.clustermap(data, method = "ward")Métrica de distancia
La métrica de distancia es la medida utilizada para calcular la distancia entre observaciones. Las métricas disponibles son: "braycurtis", "canberra", "chebyshev", "cityblock", "correlation", "cosine", "dice", "euclidean" (por defecto), "hamming", "jaccard", "jensenshannon", "kulsinski", "mahalanobis", "matching", "minkowski", "rogerstanimoto", "russellrao", "seuclidean", "sokalmichener", "sokalsneath", "sqeuclidean" y "yule". Puedes encontrar más información acerca de cada métrica en el siguiente enlace.
Métrica de Canberra
import numpy as np
import seaborn as sns
# Simulación de datos
np.random.seed(2)
data = np.random.rand(6, 6)
sns.clustermap(data, metric = "canberra")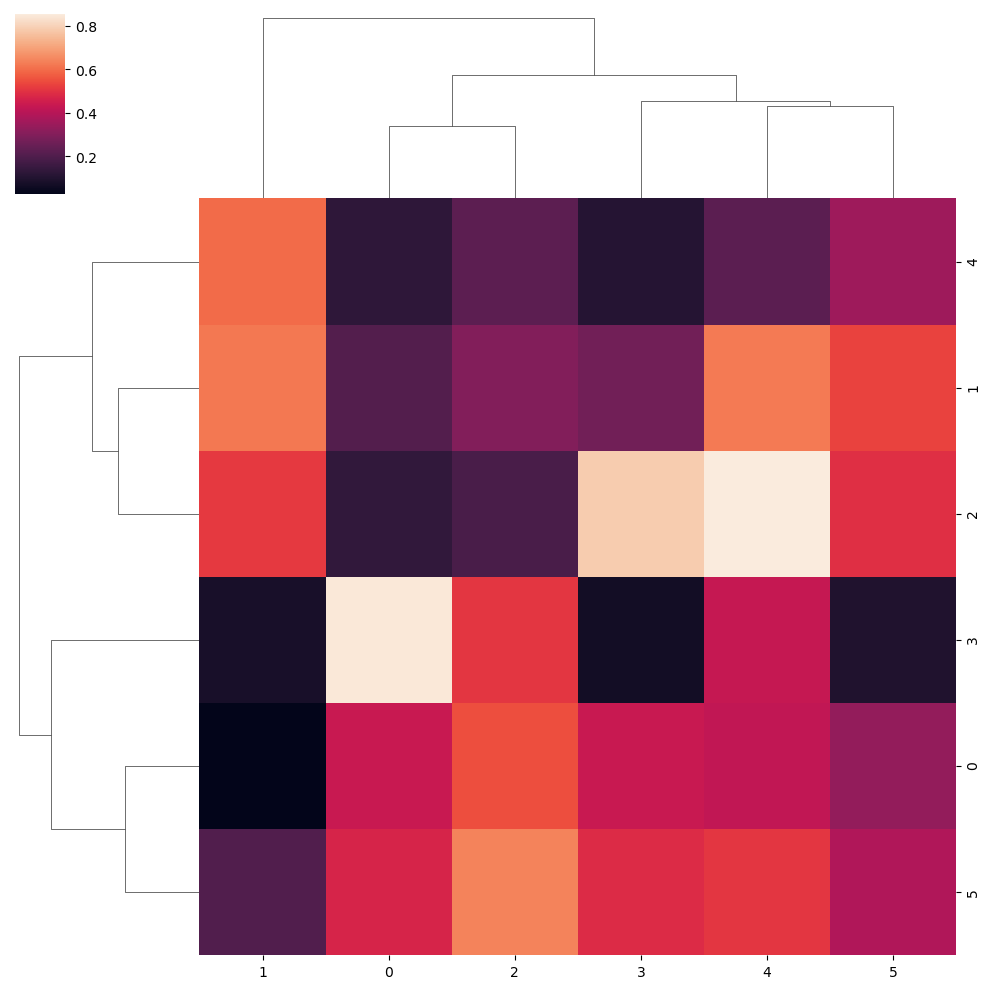
Personalización del color
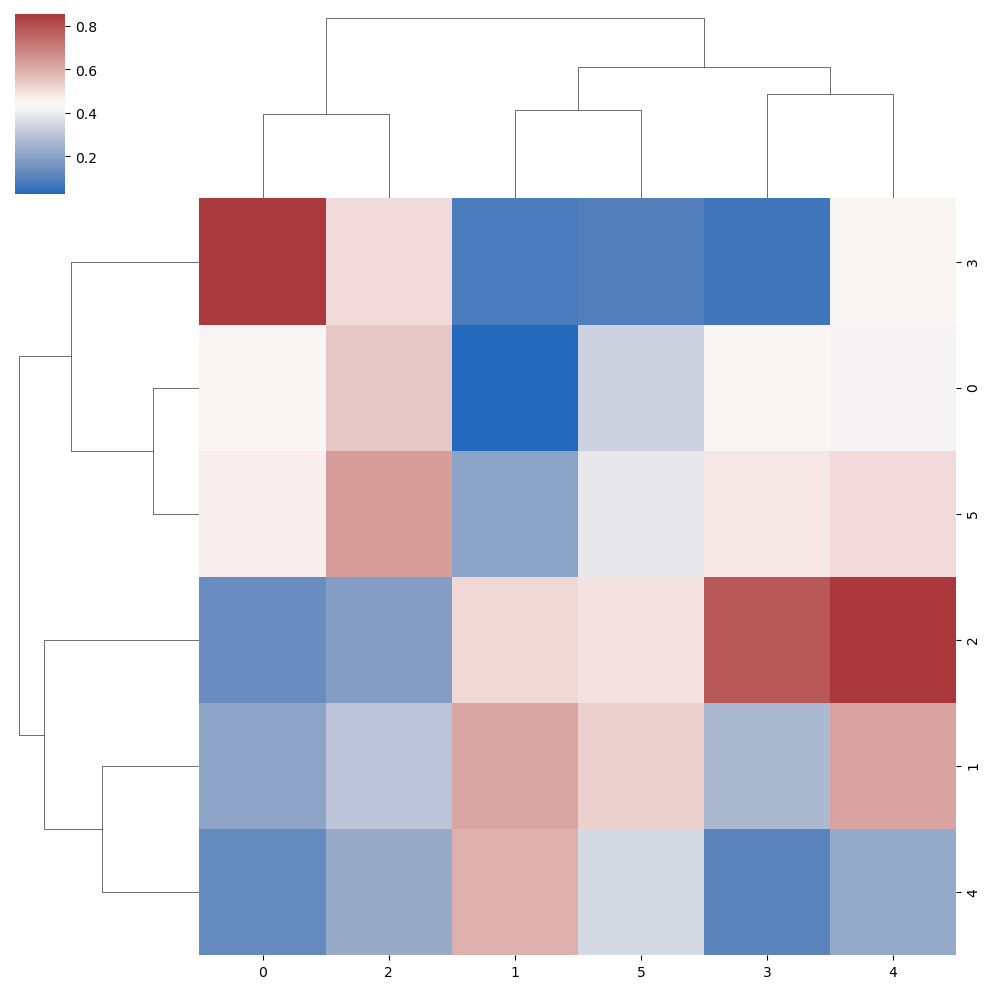
Paleta de colores
El argumento cmap se puede utilizar para cambiar la paleta de colores del mapa de calor con dendrogramas de seaborn.
import numpy as np
import seaborn as sns
# Simulación de datos
np.random.seed(2)
data = np.random.rand(6, 6)
sns.clustermap(data, cmap = "vlag")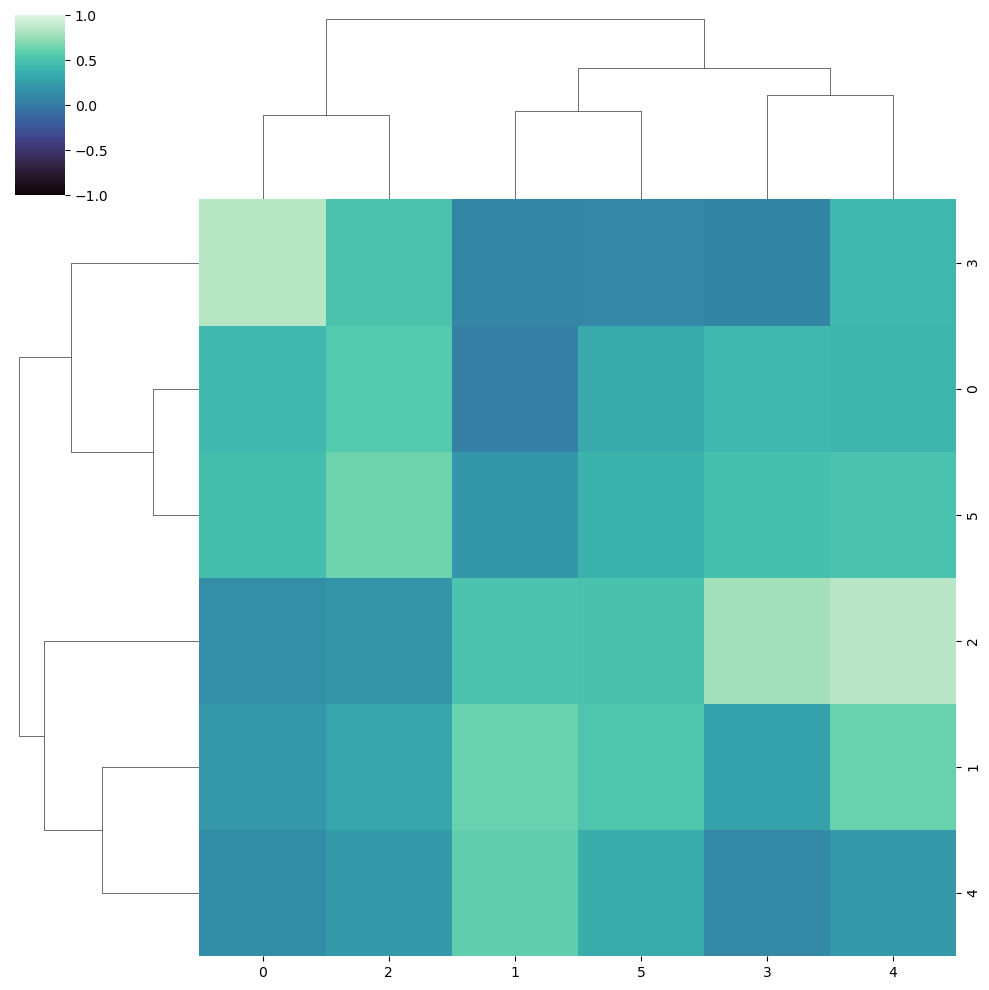
Límites de la escala de color
Ten en cuenta que puedes cambiar los límites de la escala de color con los argumentos vmin y vmax.
import numpy as np
import seaborn as sns
# Simulación de datos
np.random.seed(2)
data = np.random.rand(6, 6)
sns.clustermap(data, cmap = "mako",
vmin = -1, vmax = 1)